单细胞转录组分析不能局限在基因的差异表达,需要具体到某个生物学功能上来,才能近一步揭示生物学意义。通常进行差异基因的功能富集分析,包括GO、KEGG、REACTOME等数据库。
接下来介绍如何从Reactome 数据库下载相关基因信息
本方法参考Y叔写的两个R包,具体安装教程请自行搜索 (Bioconductor)
Step1 进入Recatome 网站搜索所需基因集
以Cell-Cell communication为例 -- https://reactome.org/PathwayBrowser/#/R-MMU-1500931&DTAB=DT
Reactome 包含多个物种,根据所需物种及时调整。
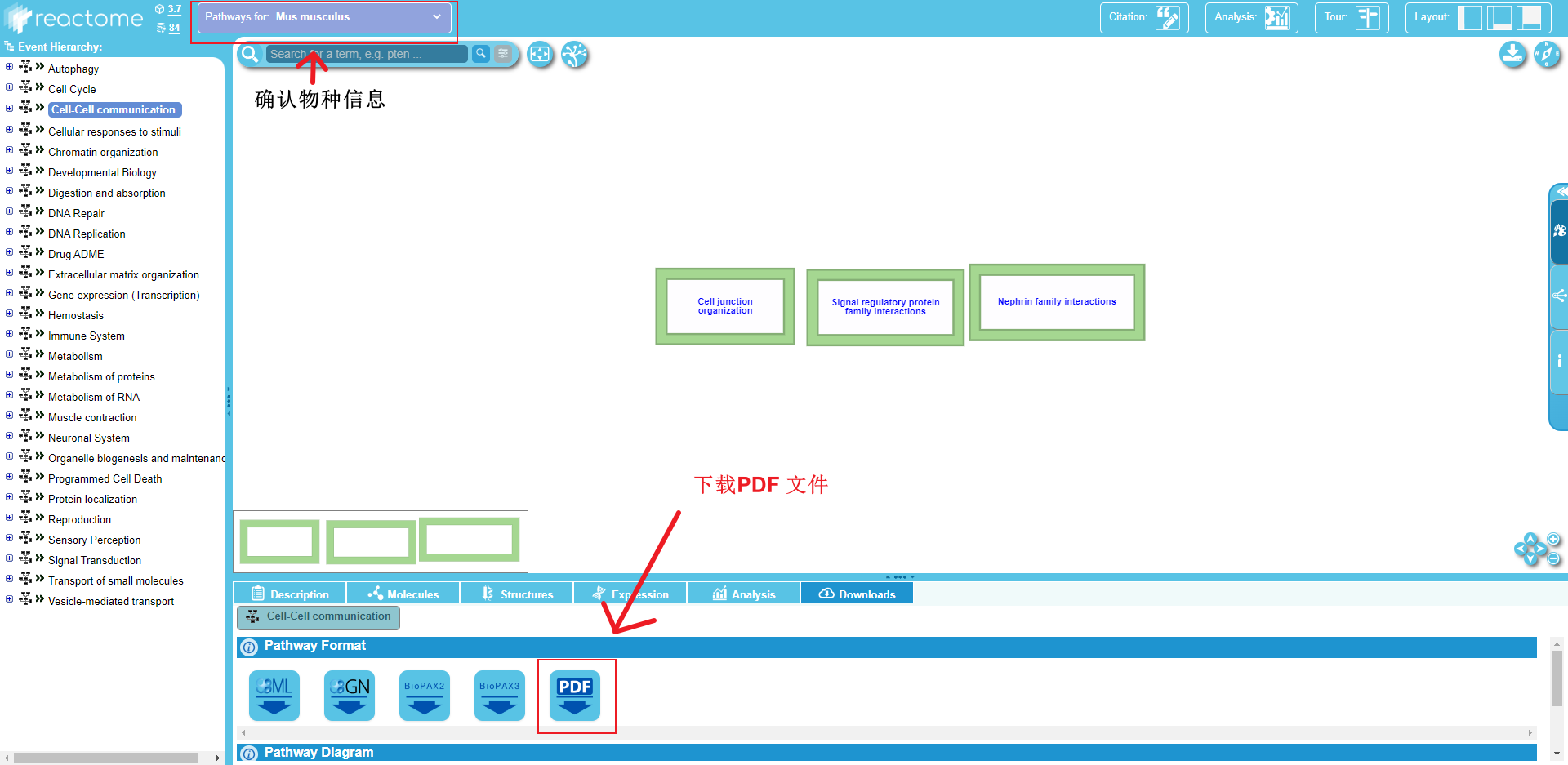
Cell-Cell Communication 相关信息
Step2 根据基因集编号进行下载
Step2.1 打开下载PDF文件
根据PDF文件中的基因集编号下载,该PDF文件中存在三个亚基因集合,分别是 Cell junction organization , Signal regulateory protein family interactions , Nephrin family interaction。如何你的课题需更准确的信息可以根据下一级基因编号下载相应数据。
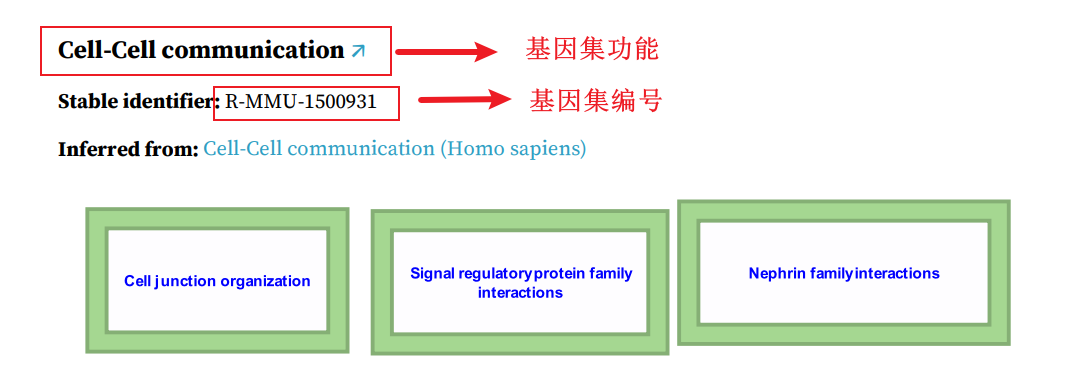
Cell-Cell Communication PDF文件相关信息
Step2.2 下载基因集
代码语言:txt
复制
library(ReactomePA)
library(reactome.db)
library(dplyr)
# Cell-Cell communication R-MMU-1500931
Cell_communication= as.list(reactomePATHID2EXTID)$ "R-MMU-1500931"
write.csv(as.data.frame(Cell_communication),'Reactome基因功能分析/mm_Cell_communication.csv')Step3 修改基因ID
由于 Reactome 存放的基因id为Enterze id,需要将其转换为常用的Symbol id
Step3.1 使用clusterProfiler 进行转换(不推荐)
丢失大量基因
代码语言:txt
复制
library("clusterProfiler")
gene.df <- bitr(genes, fromType = "ENTREZID", #fromType是指你的数据ID类型是属于哪一类的
toType = c("ENSEMBL", "SYMBOL"), #toType是指你要转换成哪种ID类型,可以写多种,也可以只写一种
OrgDb = org.Hs.eg.db)#Orgdb是指对应的注释包是哪个Step3.2 使用Ensembl数据库信息进行ID互换
Ensembl 数据库存放多种基因ID,下载到本地实现ID互换
Step3.2.1 下载相关信息
进入BioMart 选取需要的基因ID
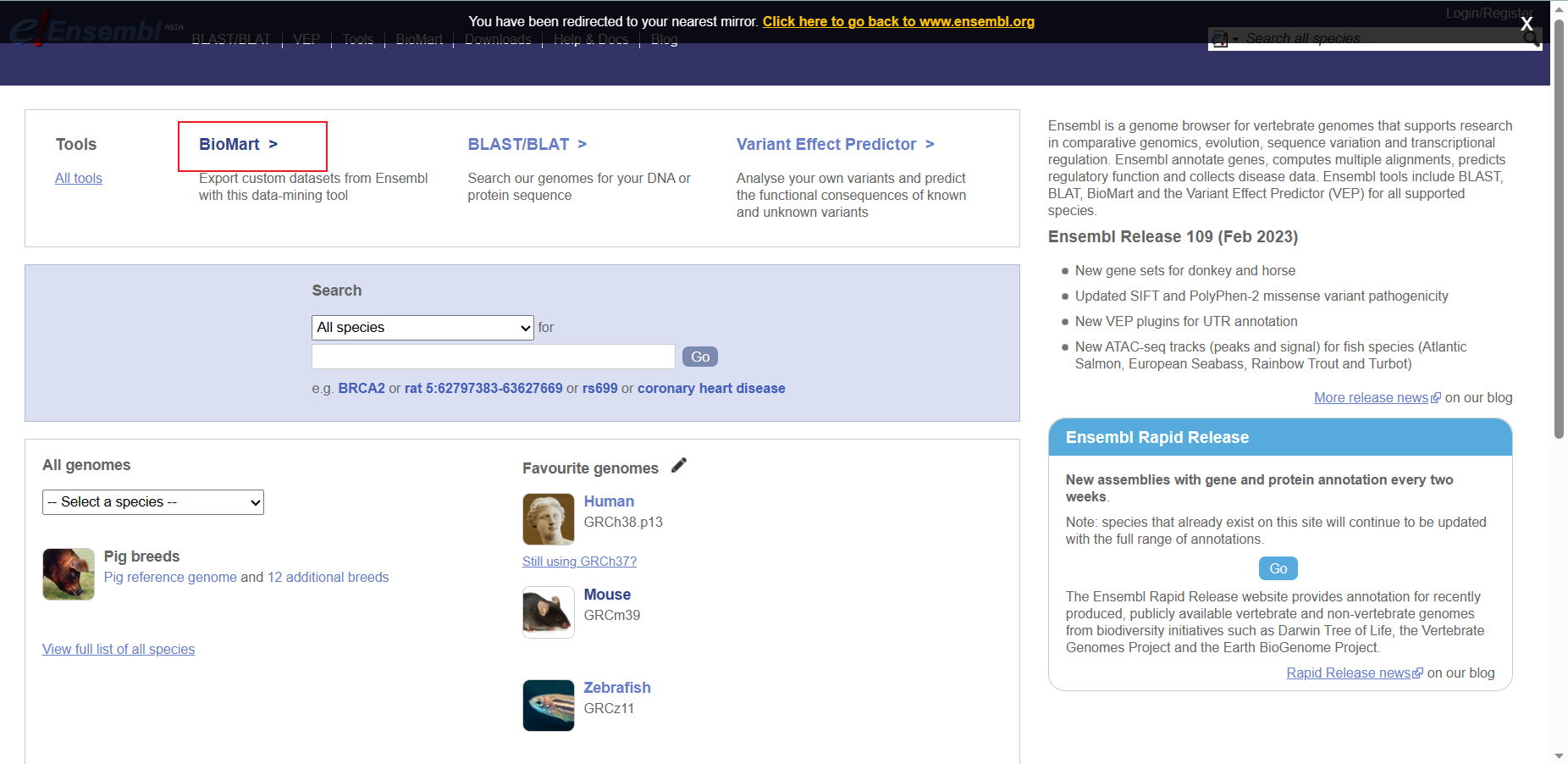
进入BioMart
选取需要的Symbol ID Entrez Id
进入Result 进行下载 得到所有信息TXT文件
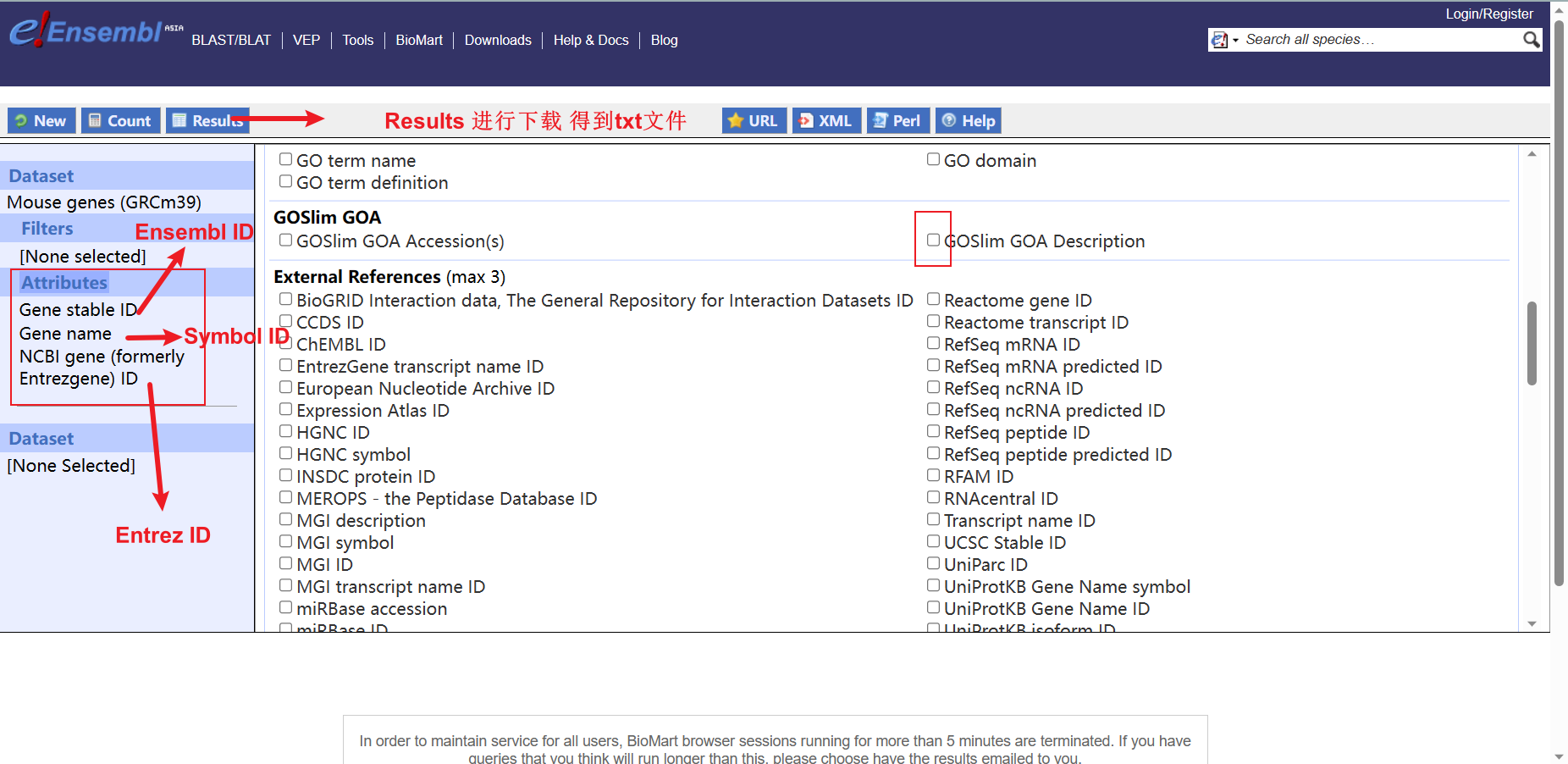
在相应的地方实现框选
Step3.2.2 进入R完成ID转换
代码语言:txt
复制
rea = read.csv('/sc-seq/database//Reactome//mouse_id exchange.txt')
cell_commun = read.csv('/sc-seq/database//Reactome//mm_Cell_communication.csv')
rea1 = rea[rea$NCBI.gene..formerly.Entrezgene..ID %in% cell_commun$Cell_communication,]
a = rea1[!duplicated(rea1$Gene.name),]
write.csv(a,'cell_communication_new.csv')后续分析就可以基于new.csv进行啦!