泛基因组分析中通常会使用orthofinder去分析基因家族,将这些基因家族分为核心和可变、私有等,然后会算不同类别的基因家族的Ka/Ks的值,主要说明的问题就是核心基因家族相对比较保守
具体怎么操作
我之前的做法是使用ParaAT这个软件去做序列比对,然后使用KaKs_Calculator计算Ka Ks的比值,整个过程还是稍微比较麻烦的
wgd这个软件有一步可以算Ks ka
使用很方便,直接修改orthofinder的输出结果,然后用wgd 的命令来算就可以
(还没有实际操作过,有空的话试试,下面的内容只是用一个基因组的cds序列做测试,学习一下软件的使用)
关于ks值和全基因组复制的关系,可以参考一下这个链接
https://www.jianshu.com/p/21e972a6e4f7
具体怎么安装和算可以参考一下这个链接
https://zhuanlan.zhihu.com/p/123875815
wgd 的github主页
https://github.com/arzwa/wgd
论文 https://academic.oup.com/bioinformatics/article/35/12/2153/5162749
每一个子命令都有很多依赖的软件,可以参考这个链接
https://wgd.readthedocs.io/en/latest/
所有的依赖都是可以用conda安装的,paml是按照wgd的github主页手动安装的,其他的都是通过conda安装
这里最开始新建conda环境python版本默认是3.12了,用3.12的python安装wgd的时候会报错,暂时不知道是什么原因,所以最开始新建环境的时候需要指定python版本,我用3.9是没问题的
conda create -n wgd python=3.9
conda activate wgd
conda install mamba
mamba install blast mcl mafft muscle prank fasttree phyml
暂时不做共线性分析,下面的依赖就先不安装了
按照wgd的github主页下载软件,setup.py里48行的sklearn需要改为scikit-learn
pip install . -i https://pypi.tuna.tsinghua.edu.cn/simple
我这里用二倍体草莓的T2T试一下
首先是运行
wgd mcl -n 48 --cds --mcl -s fv.cds -o fv.cds.out
计算kaks
wgd ksd --n_threads 48 fv.cds.out/fv.cds.blast.tsv.mcl fv.cds
fv.cds.out/fv.cds.blast.tsv.mcl 这个文件的格式是

image.png
我们把orthofinder的输出结果也整理成这种
同一个家族的基因ID放到一行,制表符分隔,orthofinder的输出结果里有这个格式的文件
计算kaks的输出
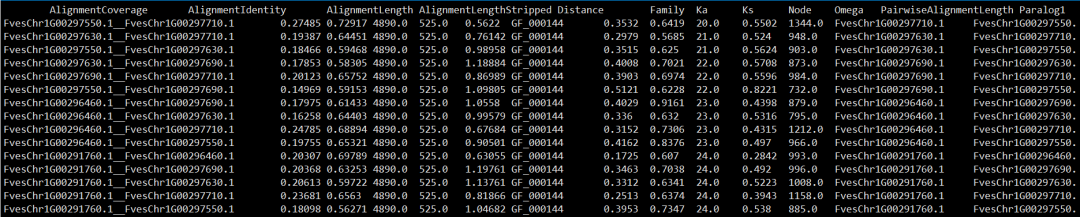
image.png
运行过程如果遇到如下报错
ImportError: cannot import name 'bool' from 'numpy'
把numpy的版本改为 1.23
pip install numpy==1.23 -i https://pypi.tuna.tsinghua.edu.cn/simple
image.png
欢迎大家关注我的公众号
小明的数据分析笔记本
小明的数据分析笔记本 公众号 主要分享:1、R语言和python做数据分析和数据可视化的简单小例子;2、园艺植物相关转录组学、基因组学、群体遗传学文献阅读笔记;3、生物信息学入门学习资料及自己的学习笔记!