欢迎关注”生信修炼手册”!
在chip_seq的实验中,由于抗体反应的敏感性,生物学重复样本的一致性很难把控。为了保证重复样本具有较好的一致性,除了在实验上保证操作流程的规范化,对于测序数据,我们也需要对其进行评估。
deeptools是一款非常好用的工具,提供了很多有用的功能,本文主要介绍如何使用deeptools来计算样本间的相关性,并进行可视化,主要分为以下两个步骤
1. 计算bam文件的coverage
对于转录组数据,通常通过样本的表达谱来计算样本间的相关性,对于chip-seq等没有明确定量结果的数据,通常的策略是将基因组划分为等长的区间,称之为bin, 计算每个区间内的覆盖度,然后通过比较不同样本间的覆盖度来计算样本相关性,借助deeptools中的multiBamSummary命令,可以实现这一功能,用法如下
代码语言:javascript
复制
multiBamSummary bins \
--bamfiles file1.bam file2.bam \
--binSize 10000 \
--numberOfProcessors 10 \
--outRawCounts results.txt \
-o results.npz \2. 可视化
上一步产生的后缀为npz的文件,通过plotCorrelation命令可以计算相关性,该命令支持spearman和pearson两种相关性分析,pearson相关系数建立在数据符合正态分布的基础上,而spearman相关系数会根据数据的排序即秩进行分析,所以会数据分布没有任何要求,但是对应的敏感性会低一点。
该命令的基本用法如下
代码语言:javascript
复制
plotCorrelation \
-in reads.npz \
--corMethod spearman \
--skipZeros \
--plotTitle "Sperman Correlation of Read Counts" \
--whatToPlot heatmap \
--colorMap RdYlBu \
--plotNumbers \
-o heatmap_SpearmanCorr.pdf \
--outFileCorMatrix SpearmanCorr_readCounts.tab输出结果示意如下
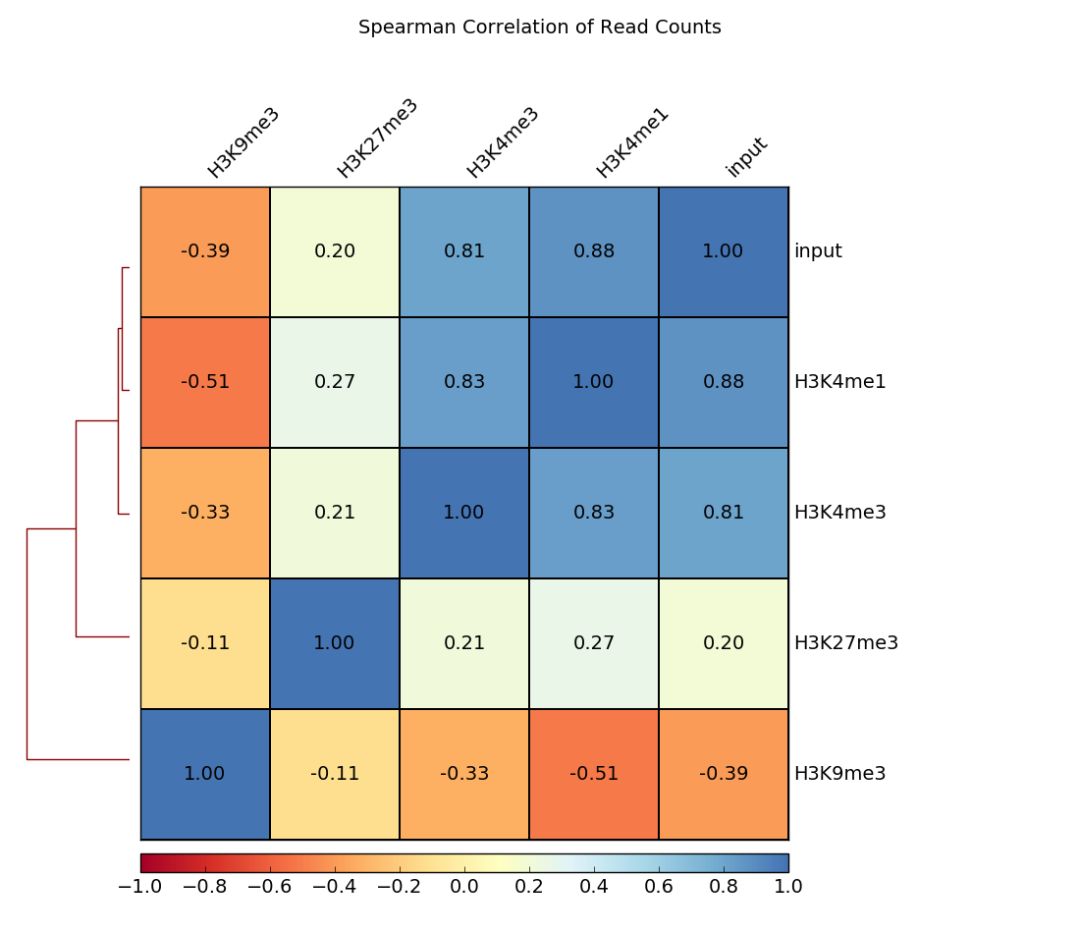
图上标记的是相关系数,除了热图之外,还支持输出两两比较的散点图,用法如下
代码语言:javascript
复制
plotCorrelation \
-in results.npz \
--corMethod pearson \
--skipZeros \
--plotTitle "Pearson Correlation of Average Scores Per Transcript" \
--whatToPlot scatterplot \
-o scatterplot_PearsonCorr.pdf \
--outFileCorMatrix PearsonCorr_bigwigScores.tab输出结果如下
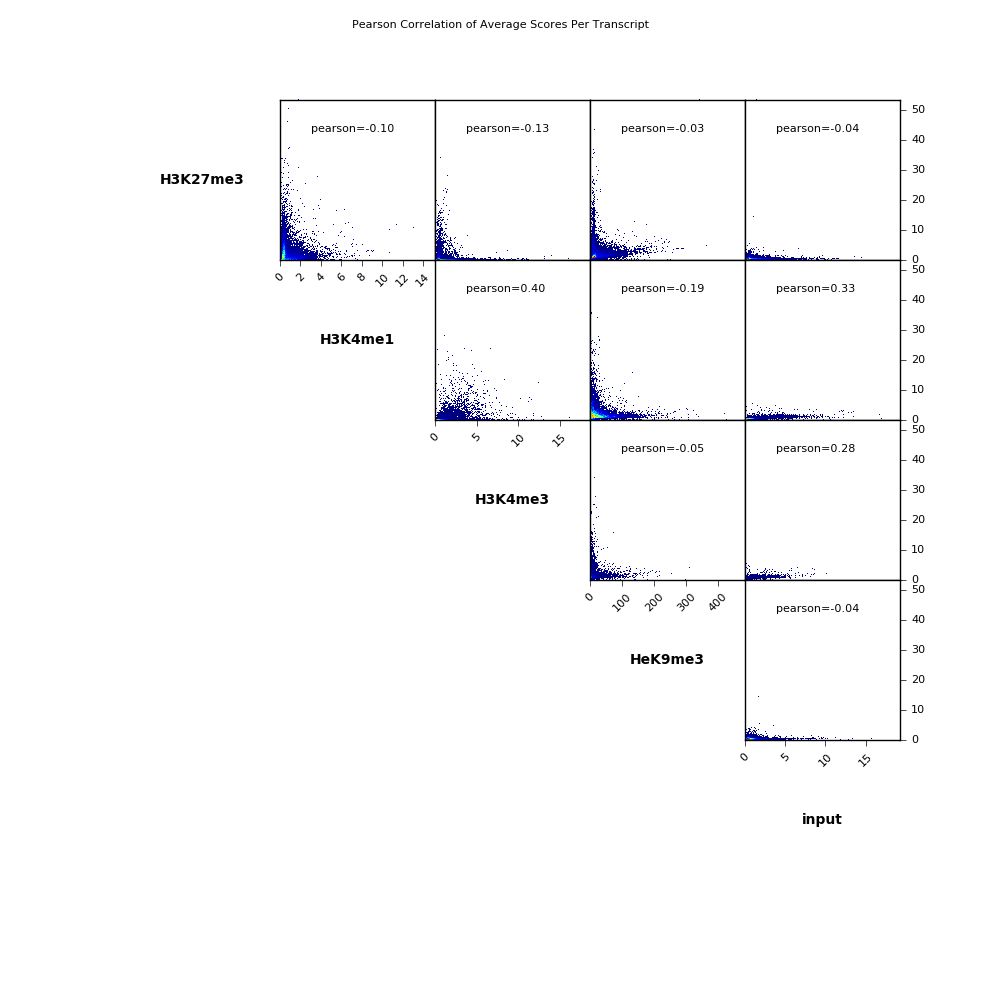
通过deepTools,可以很方便的对生物学重复的一致性进行评估。
·end·
—如果喜欢,快分享给你的朋友们吧—
扫描关注微信号,更多精彩内容等着你!