一、基因家族定义
基因家族:gene family,是指来源于同一个祖先,经过基因重复和突变而产生的一组具有序列结构与功能相似性的基因,它们编码相似的蛋白质产物。对于一个基因家族中的基因,能够编码蛋白都有同一个结构域。
维基百科:https://en.wikipedia.org/wiki/Gene_family
同源基因:来自共同祖先 DNA 序列的基因,包括了物种间的同源“ortholog”、物种内的因基因复制导致的同源"paralog"。
直系/垂直同源基因:同一祖先但后来形成了不同物种,它们之间的基因集。一般从进化上来讲,有相同的功能,但这个并不绝对,可能在新测序的物种中又会发现新的基因功能。
旁系/平行同源基因:某个特定基因组中由于基因复制产生的同源基因,直系基因在进化中一般会保持相同的功能,但是旁系基因会发生进化,可能已经有了新的功能,或者成为了假基因。
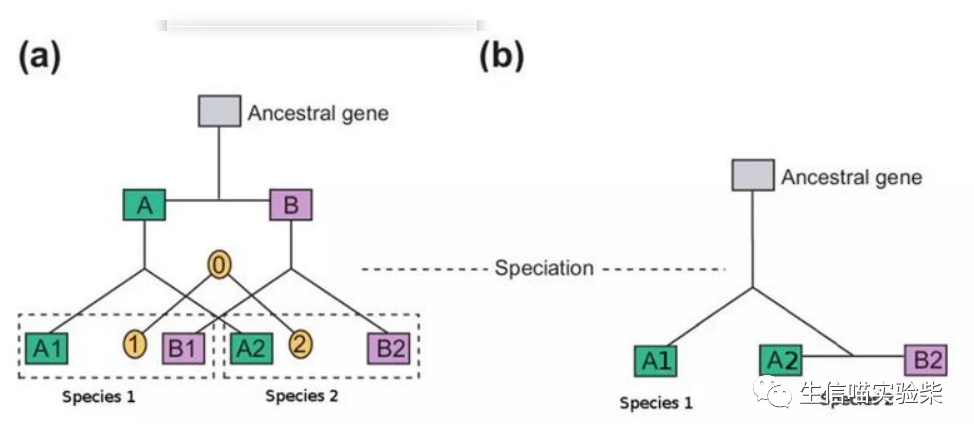
直系同源(同颜色)与旁系同源(不同颜色)
一个基因家族,一般存在于多个物种(不同物种中的叫亚家族基因),并且很多基因家族都是转录因子,可以对家族内基因启动子区域进行分析;还能找几个物种,分析同源基因的基因结构、motif 分析,找到保守的 motif;还可以结合湿实验 QRT-PCR,找出十几个或二十个基因做不同组织的表达量,看哪些基因具有较强的组织特异性表达性。
二、使用案例:水稻 Dynamin_N 基因家族
1、序列下载:下载水稻全部氨基酸序列以及 GFF 文件
https://phytozome.jgi.doe.gov/pz/portal.html
http://plants.ensembl.org/index.html
axel -n 100 http://ftp.ensemblgenomes.org/pub/plants/release-53/fasta/oryza_sativa/pep/Oryza_sativa.IRGSP-1.0.pep.all.fa.gz
axel -n 100 http://ftp.ensemblgenomes.org/pub/plants/release-53/gff3/oryza_sativa/Oryza_sativa.IRGSP-1.0.53.gff3.gz2、下载水稻 Dynamin_N 家族 hmm 文件
http://pfam.xfam.org/family/PF00350#curationBlock
wget http://pfam.xfam.org/family/PF00350/hmm
mv hmm Dynamin_N.hmm3、hmmersearch 比对
#hmmersearch
hmmsearch -o hmmer.out --domE 1E-5 -E 1E-5 Dynamin_N.hmm
Oryza_sativa.IRGSP-1.0.pep.all.fa
#复制比对上的基因 ID,保存到 ids.txt 文件中
#根据比对上的 ID 提取序列
samtools faidx Oryza_sativa.IRGSP-1.0.pep.all.fa
cat ids.txt |xargs -n 1 samtools faidx
Oryza_sativa.IRGSP-1.0.pep.all.fa >Oryza_family.fa三、motif 特征分析
Motif 是在生物学中是一个基于数据的数学统计模型,典型的是一段 sequence 也可以是一个结构。如转录因子倾向于结合某些特定的序列。
在线分析网站:http://meme-suite.org/tools/meme
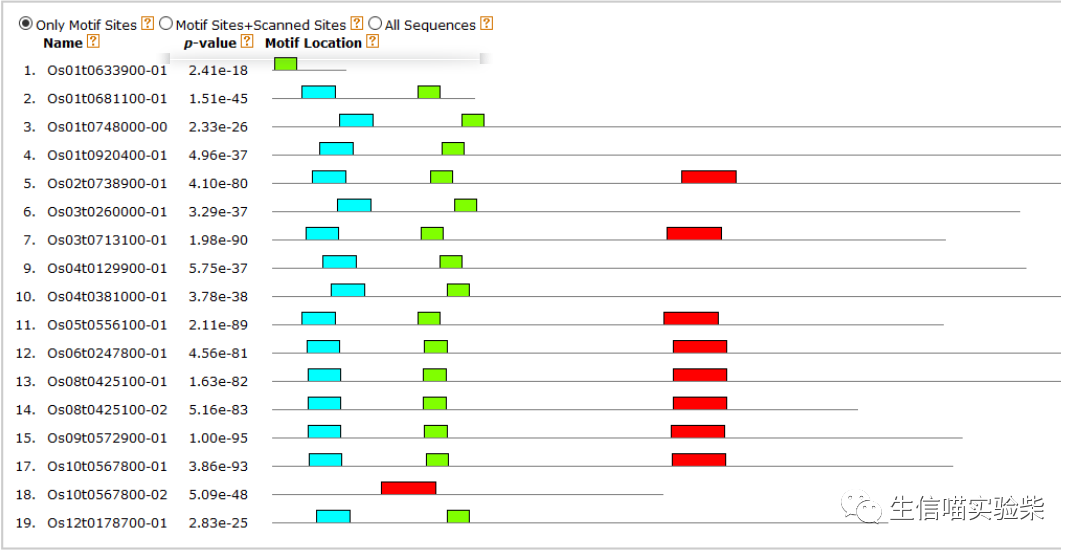
基因功能结构域
四、基因结构分析
GSDS: Gene Structure Display Server
for i in `cat ids.txt`;do grep "$i" Oryza_sativa.IRGSP-1.0.53.gff3 >>Oryza_21.gff;done;在线分析网站:http://gsds.gao-lab.org/
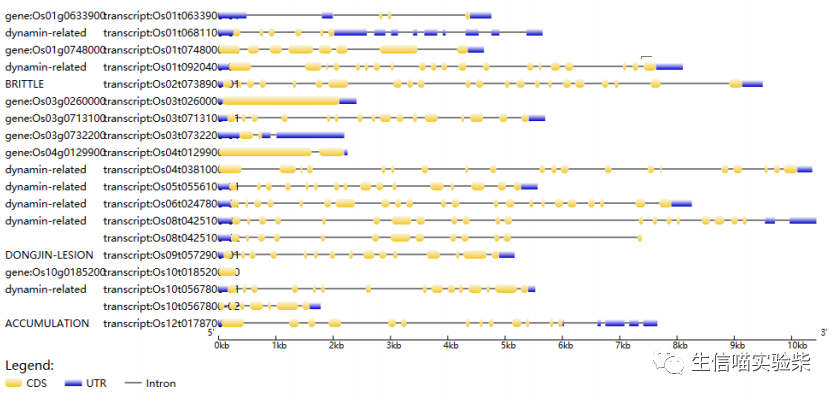
基因结构分析可视化
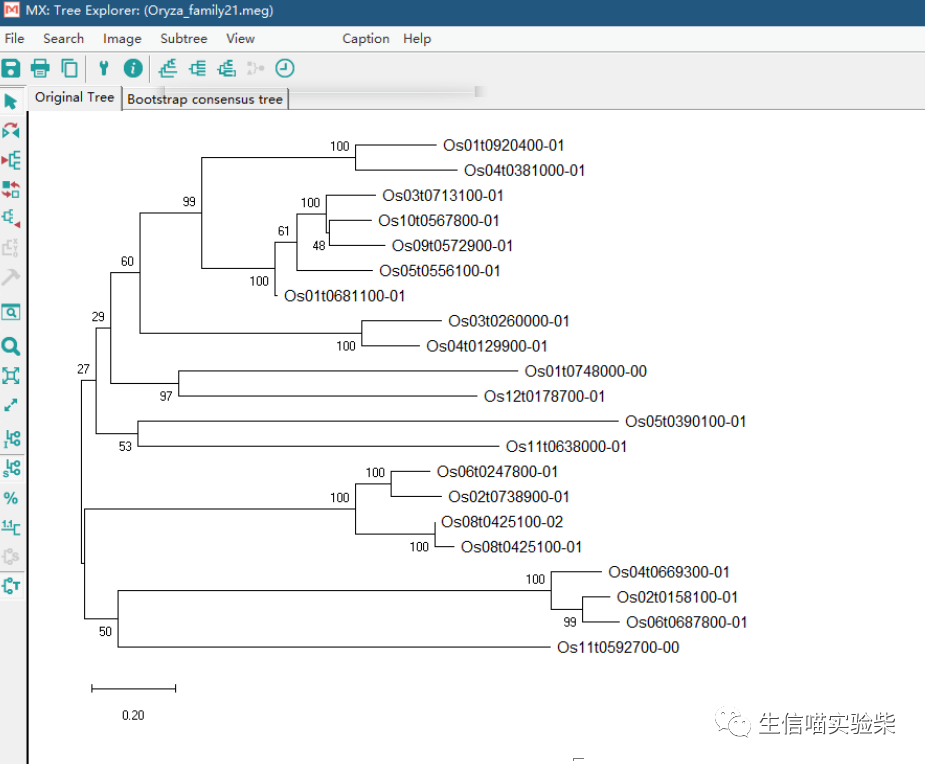
五、系统发育树构建
利用 megax 对水稻中整个 Dynamin 基因构建系统发育树。
写在最后:有时间我们会努力更新的。大家互动交流可以前去论坛,地址在下面,复制去浏览器即可访问,弥补下公众号没有留言功能的缺憾。原地址暂未启用(bioinfoer.com)。
sx.voiceclouds.cn有些板块也可以预设为大家日常趣事的分享等,欢迎大家来提建议。