简明易懂爬虫教程
后半部分需要用到selenium但是目前电脑的chrome版本过高,就先不继续后半部分的代码编写
首先上网站:
https://toolkit.tuebingen.mpg.de/然后使用 HHblits进行同源序列的搜索:
HHblits是一种用于远程蛋白质序列比对的工具。它是基于隐马尔可夫模型(HMM)的迭代比对方法,用于识别结构和功能相关的蛋白质序列之间的相似性。HHblits的工作流程如下:
构建初始HMM库:使用已知的蛋白质序列和结构信息构建初始的HMM库。
迭代比对:将待比对蛋白质序列与HMM库进行比对,得到一组相关的序列。接着,在每次迭代中,使用这些相关序列构建更新的HMM库,并与待比对序列进行进一步的比对。迭代过程将重复多次,逐步提高比对的准确性和覆盖度。
终止迭代:在达到预定的迭代次数或收敛条件后,停止迭代过程,并输出最终的比对结果。
HHblits主要用于寻找远程同源蛋白质序列,即那些在传统序列比对方法中难以检测到的相似性。它在蛋白质结构预测、功能注释和进化分析等领域具有广泛的应用。HHblits具有以下特点:
迭代过程:通过多次迭代比对和更新HMM库,提高了比对的准确性和灵敏度。
丰富的特征集:HHblits使用了丰富的特征集,包括氨基酸残基的保守性、序列复杂性和二级结构预测等信息,从而提高了比对的质量。
高效的算法:HHblits通过使用索引和压缩技术,提高了比对的速度和效率。
内嵌的结构信息:HHblits结合了蛋白质序列和结构信息,可以更好地捕捉到序列和结构之间的关联性。
总体而言,HHblits是一种强大的工具,可用于发现远程同源蛋白质序列之间的相似性,为蛋白质结构和功能的研究提供重要支持。
搜索完的页面就是这个样子:
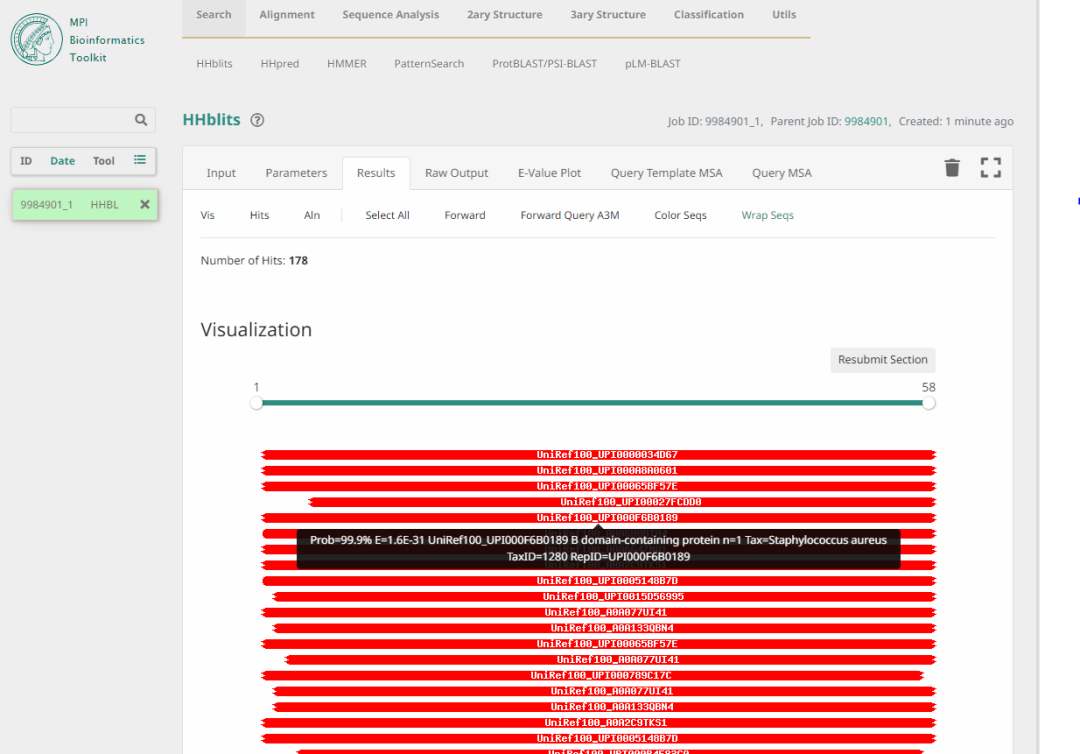
但是今天我们不学网站怎么使用,主要看如何爬取数据
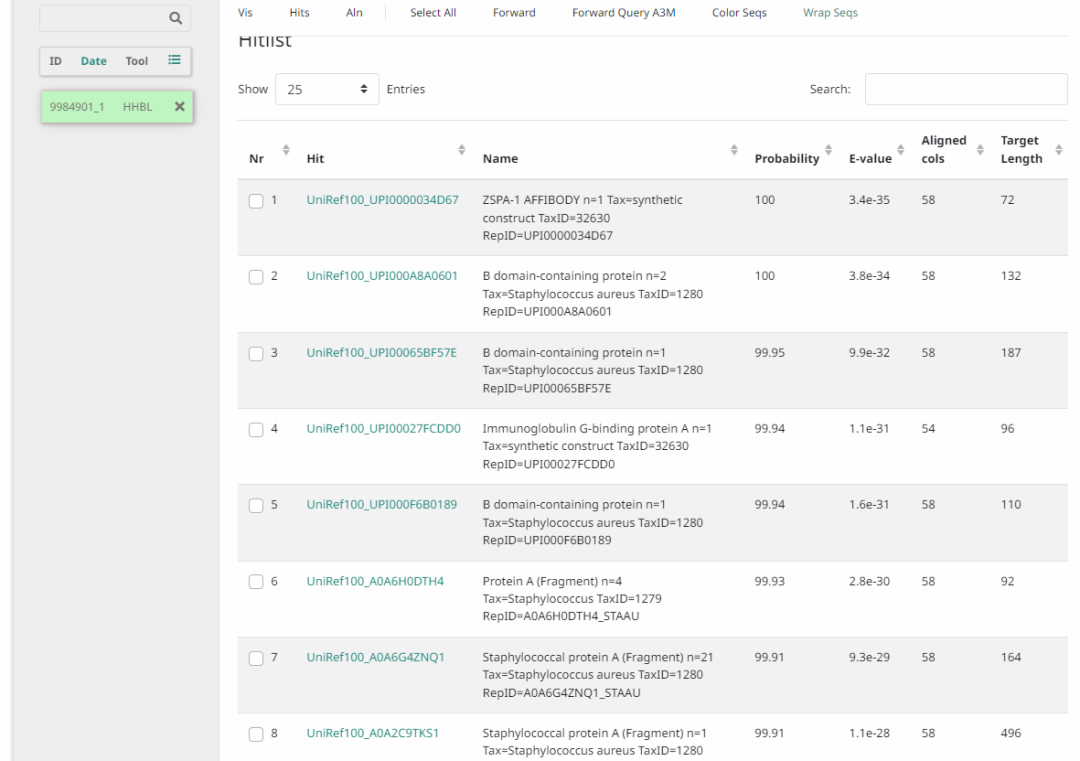
这一块是我们的结果区域
然后研究了请求过程之后发现每次搜索同源序列都会形成一个job id
然后job id是固定的也是唯一的,所以我们只需要针对这一个job id的网址进行爬取即可
简单说一下要求,今天的要求是爬取长度是100以内的数据即可:

然后直接上代码:
import requests import re from selenium import webdriver #同源序列的搜索 和数据的下载task_url="https://toolkit.tuebingen.mpg.de/api/jobs/你的jobid/results/hits/?start=0&end=250&filter=&sortBy=&desc=false"
headers={
'User-Agent':'Mozilla/5.0 (Windows NT 10.0; Win64; x64) AppleWebKit/537.36 (KHTML, like Gecko) Chrome/117.0.0.0 Safari/537.36'}
response=requests.get(headers=headers,url=task_url)
hits_list=response.json()['hits']
hits_url_list=[]
for i in hits_list:
if i['templateRef']<100 or i['templateRef']==100:
hits_url_list.append(i['acc'])pattern = r"<a href='(.*?)' target='_blank'>"
sequence_url=[]
for ul in hits_url_list:
match=re.search(pattern,ul)
se_url=match.group(1)
sequence_url.append(se_url)
with open('sequece.txt','a') as f:
f.write(se_url+"\n")
然后最后可以保存到本地的txt中 就可以查看符合要求的序列了:

随便点开一个链接举个例子 ,这个里面就是我们想要的序列:
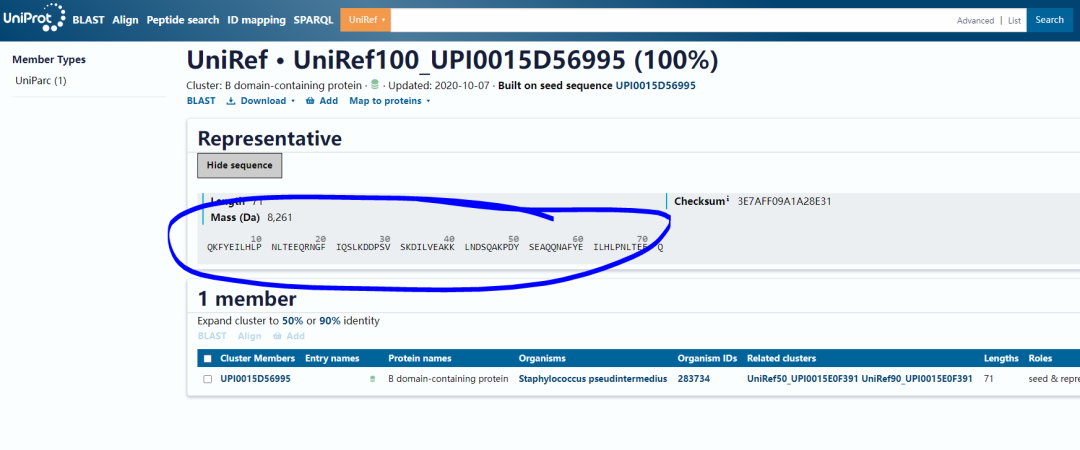
但是这里有一个js的小按钮,所以如何爬取完整的序列数据,我们将在下一篇内容继续介绍。
关于同源序列:
同源序列是指在进化过程中具有共同祖先的蛋白质序列。这些序列之间存在相似性,可以通过比较它们的氨基酸组成、序列特征和功能来推断它们的相似性和关系。同源序列通常可以分为以下几类:
同族序列(Homologous Sequences):同族序列是指具有共同祖先并具有相似结构和功能的蛋白质序列。它们通过基因复制、基因重组或基因转座等进化事件产生。同族序列通常具有相似的三级结构和保守的功能域,常常拥有相似的生物学功能。
异源同源序列(Orthologous Sequences):异源同源序列是指在不同物种中具有相同功能的蛋白质序列。这些序列源于共同的祖先基因,并在物种分化过程中保留了相似的功能。由于异源同源序列具有相似的功能,它们在进化和生物学功能的研究中具有重要的意义。
保守序列(Conserved Sequences):保守序列是指在不同物种或同一物种中具有高度保守性的蛋白质序列。这些序列在进化过程中经过选择压力的筛选,因为它们具有重要的生物学功能和结构特征。保守序列通常用于推断蛋白质的结构、功能和进化关系。
同源序列的研究对于理解蛋白质的结构、功能和进化具有重要意义。通过比较同源序列,可以推断蛋白质的结构和功能,预测未知序列的特性,并研究蛋白质家族的进化历史。同源序列比对和分析工具,如BLAST、HHblits和Phylogenetic Tree等,被广泛应用于同源序列的研究和分析。