《线性模型在动物育种值预测中的应用》 第二章:亲属间的遗传协方差,P19
1, 概念定义
近交系数: 近交系数(inbreeding coefficient)是指根据近亲交配的世代数,将基因的纯化程度用百分数来表示即为近交系数,也指个体由于近交而造成异质基因减少时,同质基因或纯合子所占的百分比也叫近交系数,个体中两个亲本的共祖系数。
亲缘系数: 是指两个个体间加性基因效应间的相关.
两者的区别和联系:
- 近交系数是个体的值
- 亲缘系数是两个个体之间的值
两者的计算方法:
- 可以使用通径分析的方法进行计算
- 也可以采用由系谱构建亲缘关系A矩阵的形式进行计算, 这种方法在数据比较大时更为方便
2, 系谱数据
这里我们模拟了四个个体的系谱关系, 想要计算一下每个个体的近交系数, 以及个体间的亲缘系数, 使用R语言实现.
ped <- data.frame(ID=c(3,4,5,6),Sire=c(1,1,4,5),Dam=c(2,NA,3,2))
pedID | Sire | Dam |
|---|---|---|
3 | 1 | 2 |
4 | 1 | NA |
5 | 4 | 3 |
6 | 5 | 2 |
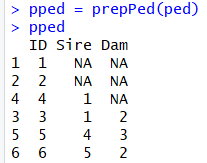
首先, 我们看到个体1和2的亲本未知, 所以我们先将系谱补充完整, 使用nadiv的prepPed函数.
library(nadiv)
pped = prepPed(ped)
pped完整的系谱如下, NA表示亲本未知.
3, 计算亲缘关系A矩阵
as.matrix(makeA(pped))这里我们使用makeA函数, A矩阵如下:
1 | 2 | 3 | 4 | 5 | 6 | |
|---|---|---|---|---|---|---|
1 | 1.00 | 0.000 | 0.5000 | 0.5000 | 0.5000 | 0.2500 |
2 | 0.00 | 1.000 | 0.5000 | 0.0000 | 0.2500 | 0.6250 |
3 | 0.50 | 0.500 | 1.0000 | 0.2500 | 0.6250 | 0.5625 |
4 | 0.50 | 0.000 | 0.2500 | 1.0000 | 0.6250 | 0.3125 |
5 | 0.50 | 0.250 | 0.6250 | 0.6250 | 1.1250 | 0.6875 |
6 | 0.25 | 0.625 | 0.5625 | 0.3125 | 0.6875 | 1.1250 |
4, 计算近交系数
用亲缘关系矩阵A的对角线-1,即是个体的近交系数
diag(A)-1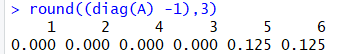
可以看出, 1,2,4,3的近交系数为0. 个体5和6的近交系数为0.125.
5, 计算亲缘系数
根据计算的亲缘关系A矩阵,这个矩阵时个体间的方差协方差矩阵, 对角线为每个个体的方差, 非对角线为个体间的协方差. 公式为:
rij = cov(i,j)/sqrt(var(i)*var(j))
因此如果我们要计算个体1和2的亲缘系数, 可以用A12/(sqrt(A11*A22)) = 0/sqrt(1*1) = 0, 因此个体1和2 的亲缘系数为0.
因为共有6个个体, 1和2的亲缘系数 = 2和1的亲缘系数, 因此他们之间的亲缘系数一共有6*5/2 = 15个. 这里我们都计算, 共有36行.
第一种方案:
n = dim(A)[1] #1
id = row.names(A) #2
mat = matrix(0,n,n) #3
for(i in 1:n){ #4
for(j in i:n){
mat[i,j] = A[i,j]/(sqrt(A[i,i]*A[j,j]))
mat[j,i] = mat[i,j]
}
}
mat #5
re = data.frame(ID1= rep(id,n),ID2=rep(id,each = n),y = round(as.vector(mat))) #6
re#7这里我们的编程思路如下:
#1 计算出矩阵的行, 确定循环数
#2 计算出个体的ID名在矩阵中的顺序, 因为有些ID可能是字符或者没有顺序, 主要用于后面的个体编号的确定
#3 为了计算更快, 我们生成一个6*6的矩阵
#4 写一个循环, 因为矩阵时对称的, 所以我再第二个for循环时从i开始, 而不是从1开始, 后面mat[j,i] = mat[i,j]再赋值, 这样更快.
#5 生成的mat矩阵查看
#6 根据ID生成两列, 表示他们之间的亲缘系数, 这个和矩阵变为向量后一一对应.
#7 查看结果
结果如下:
ID1 | ID2 | r |
|---|---|---|
1 | 1 | 1.0000 |
1 | 2 | 0.0000 |
1 | 3 | 0.5000 |
1 | 4 | 0.5000 |
1 | 5 | 0.4714 |
1 | 6 | 0.2357 |
2 | 1 | 0.0000 |
2 | 2 | 1.0000 |
2 | 3 | 0.5000 |
2 | 4 | 0.0000 |
2 | 5 | 0.2357 |
2 | 6 | 0.5893 |
3 | 1 | 0.5000 |
3 | 2 | 0.5000 |
3 | 3 | 1.0000 |
3 | 4 | 0.2500 |
3 | 5 | 0.5893 |
3 | 6 | 0.5303 |
4 | 1 | 0.5000 |
4 | 2 | 0.0000 |
4 | 3 | 0.2500 |
4 | 4 | 1.0000 |
4 | 5 | 0.5893 |
4 | 6 | 0.2946 |
5 | 1 | 0.4714 |
5 | 2 | 0.2357 |
5 | 3 | 0.5893 |
5 | 4 | 0.5893 |
5 | 5 | 1.0000 |
5 | 6 | 0.6111 |
6 | 1 | 0.2357 |
6 | 2 | 0.5893 |
6 | 3 | 0.5303 |
6 | 4 | 0.2946 |
6 | 5 | 0.6111 |
6 | 6 | 1.0000 |
第二种方案:
这里也可以用我写的learnasreml包的: mat_2_coefficient函数, 更方便.
library(learnasreml)
re2 = mat_2_coefficient(mat)
head(re2)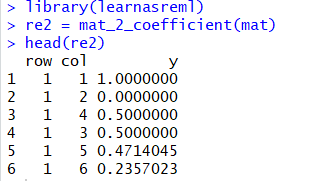
结果和上面是一致的.
所有代码汇总如下:
ped <- data.frame(ID=c(3,4,5,6),Sire=c(1,1,4,5),Dam=c(2,NA,3,2)) pedlibrary(nadiv)
pped = prepPed(ped)
ppedA = as.matrix(makeA(pped))
round((diag(A) -1),3)
A
n = dim(A)[1]
id = row.names(A)
mat = matrix(0,n,n)
for(i in 1:n){
for(j in i:n){
mat[i,j] = A[i,j]/(sqrt(A[i,i]*A[j,j]))
mat[j,i] = mat[i,j]
}
}
mat
re = data.frame(row = rep(id,n),col=rep(id,each = n),y = round(as.vector(mat)))
re
library(learnasreml)
re2 = mat_2_coefficient(mat)
head(re2)
希望可以帮到你.
转发朋友圈是最大的支持.