近年来,研究细胞与细胞相互作用(CCIs) 的工具经历了显著的多样化。2024年1月,《Nature Reviews Genetics》发表综述,探讨细胞-细胞相互作用研究的最新进展,并重点介绍新一代工具的多样化。

* 由配体-受体相互作用(LRI)介导、专门用于传递信号的细胞相互作用也被称为细胞-细胞通讯(CCC),但为简单起见,本文作者将这一特定亚型简称为 CCIs。
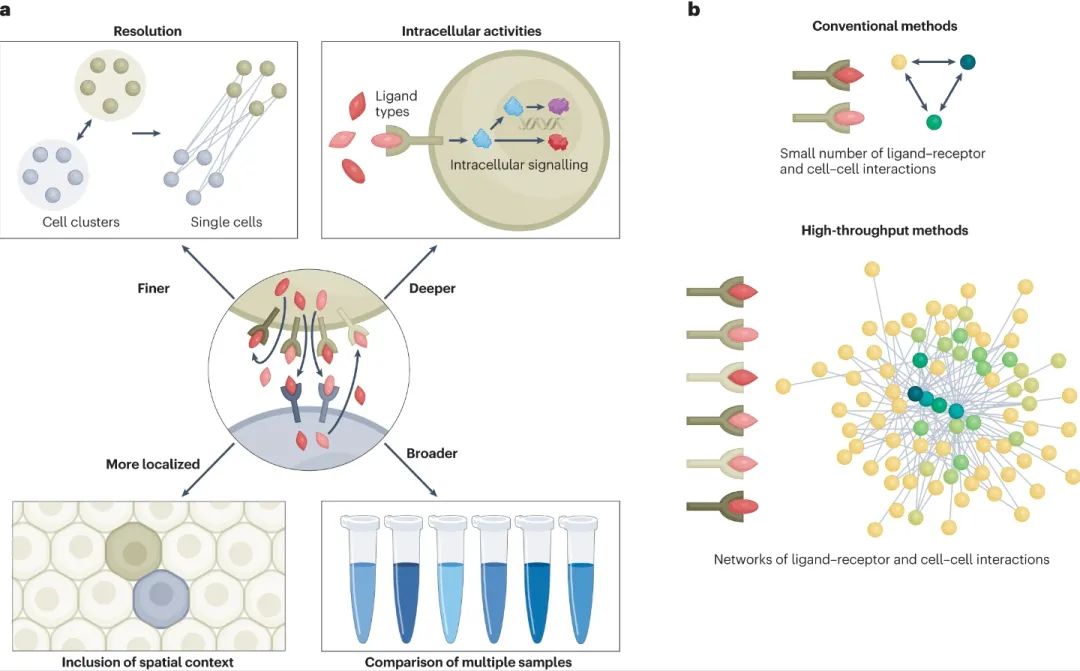
细胞-细胞相互作用研究的方法学进展
新一代计算工具
各种核心工具(如CellPhoneDB和CellChat等),一般基于配体和受体的表达,通过核心评分函数(如表达平均值、表达产物、表达相关性、基于表达的Hill函数和差异组合)推断CCIs。用于推断CCIs的计算工具是从核心工具的“根”演变而来的。从这个“根”开始,方法变得更加专门化,以应对特定的问题(单细胞、空间组等)。
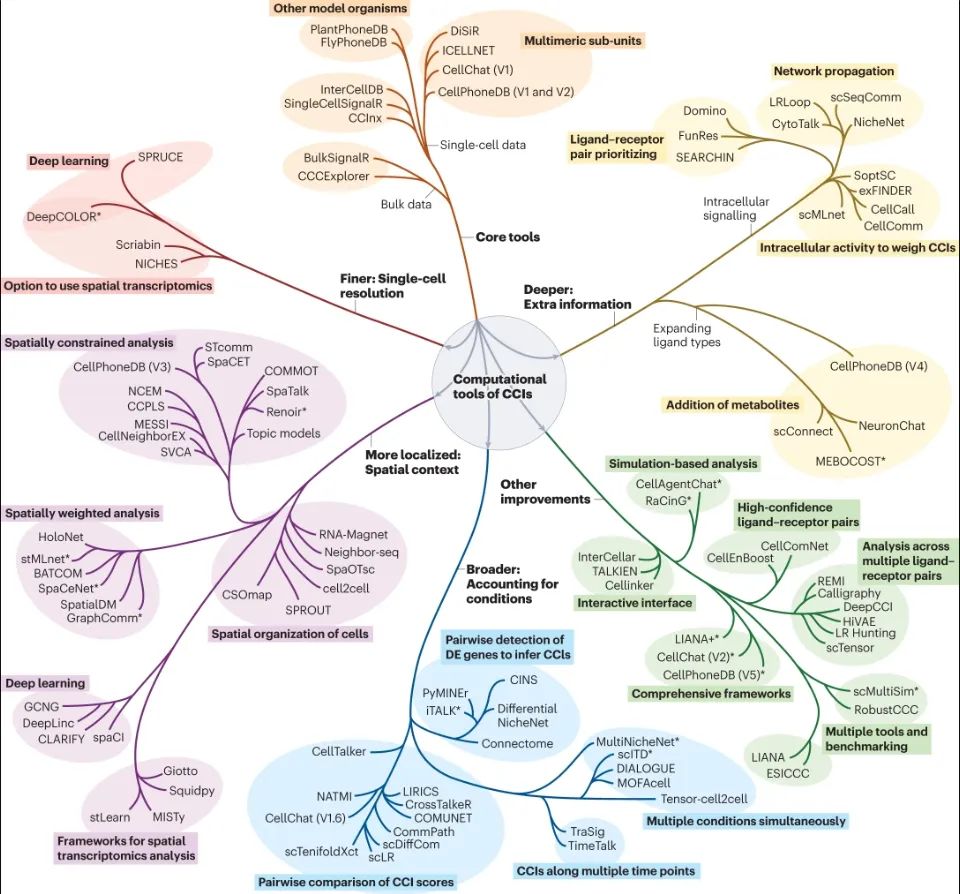
推断细胞-细胞相互作用的计算工具的系统发育树
获得全单细胞分辨率的见解:最近的方法可以以真正的单细胞分辨率处理这些数据,推断出成对的单个细胞之间的通讯,如SoptSC、NICCHES、Scriabin、SPRUCE、DeepCOLOR等。
细胞空间背景化:查每个细胞空间背景的新 CCIs工具将更清晰地解读复杂组织中具有生物意义的通讯,如Giotto、Squidpy、SVCA 、SpaOTsc、COMMOT、stMLnet、DeepLinc、Neighbour-seq、CSOmap等。
观察细胞内活动:基于转录组学的工具通常仅限于简单地推断蛋白质配体,系统生物学途径分析方法可以加深对细胞内过程的了解,以推断其他类别配体的丰度和活性,从而扩大CCIs工具的能力,如MEBOCOST、Domino、CellComm、exFINDER等。
考虑多种情况:在CCIs研究中还需要考虑细胞和LRIs所有组合的表型关联,如Connectome、CellChat、scDiffCom、 scTenifoldXct、sciTD、DIALOGUE、 MOFAcell、Tensor-cell2cell、 TraSig、 TimeTalk等。
更多其他提升:交互友好(InterCellar、Cellinker、 TALKIEN) ;LIANA建立了一个用于实施多种工具方法的平台,从而能够进行比较分析;Calligraphy使用通讯基因共表达网络来识别信号基因模块等。
新一代实验工具
新一代实验方法的出现提高了CCIs测量的通量。这些方法可以同时测量许多LRIs和细胞对,许多方法还将实验测量的相互作用与下游测序结合起来。
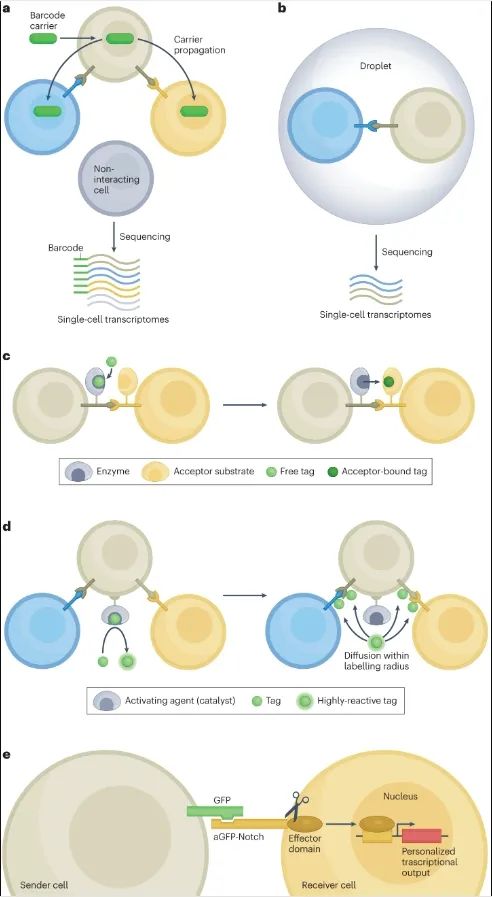
研究细胞-细胞相互作用的新一代实验方法
基于测序的方法

基于邻近标记的方法
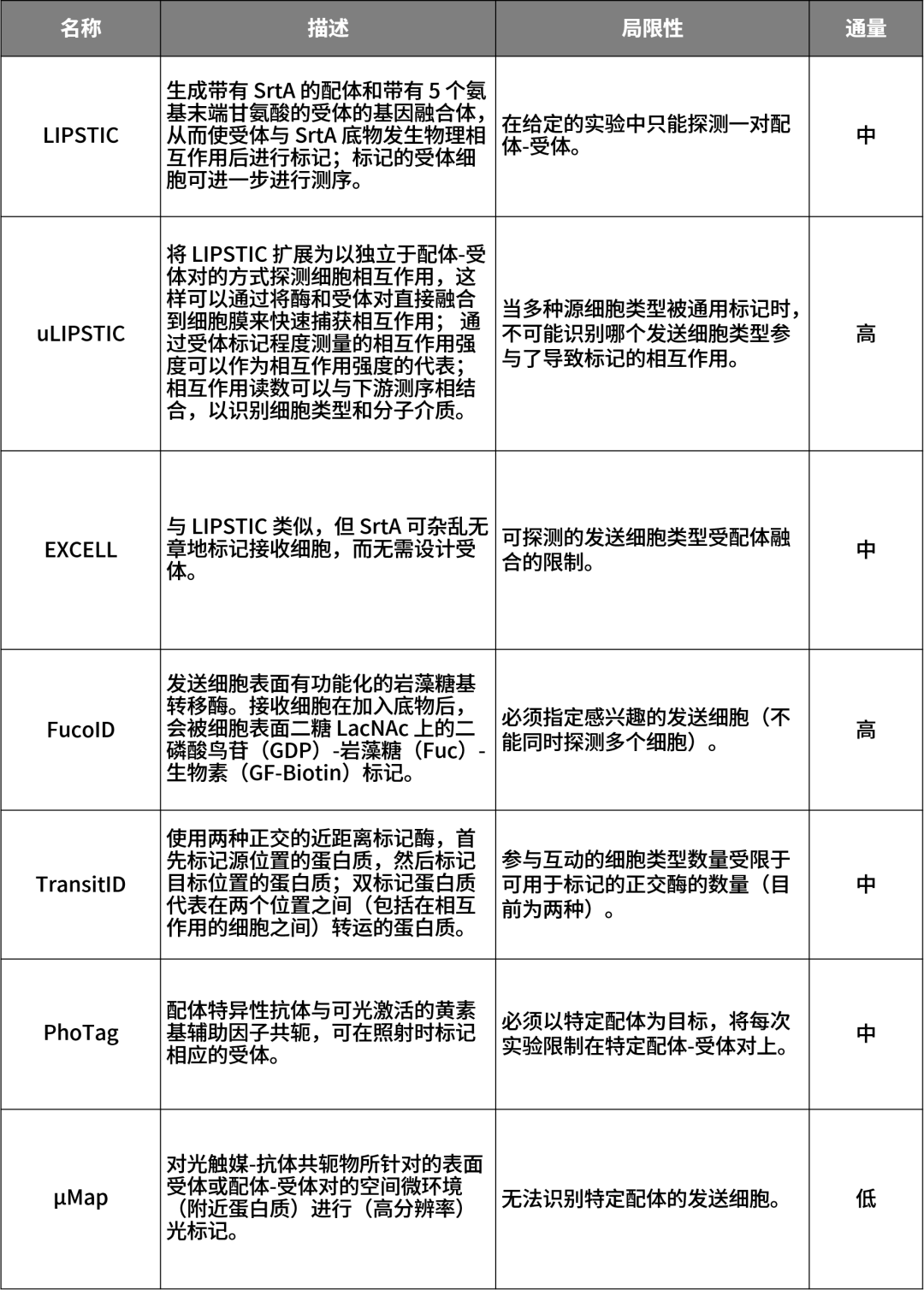
基于合成回路的方法

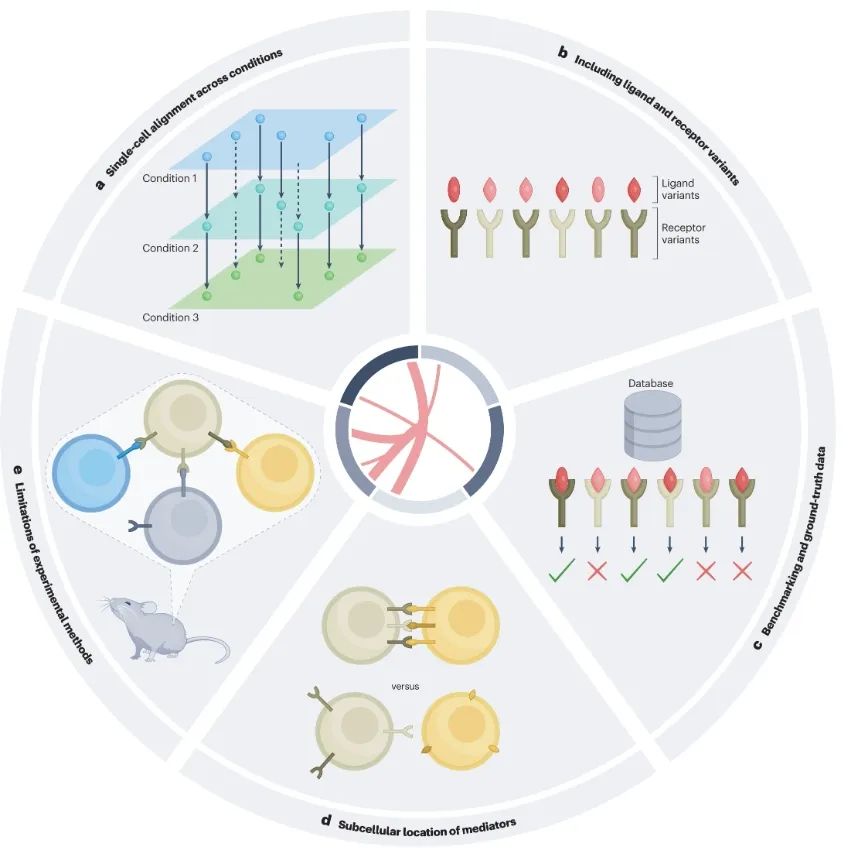
加强细胞-细胞相互作用研究方法的挑战与机遇
以单细胞分辨率进行跨条件的细胞-细胞相互作用(CCI)比较存在挑战:样本细胞数量的差异可能与某些算法不兼容,这意味着可能需要包括数据插补,尤其是在单细胞分辨率下,例如DURIAN、 CellChat (V2)、CellPhoneDB (V5)和LIANA+等算法通过不同策略改善此挑战。
构建LRIs数据库是研究CCIs的关键:这一步骤涉及一些基本方面,如关注高可信度LRIs,包括蛋白质亚基、激活剂、抑制剂和/或竞争对手,以考虑LRIs的信号活性,并结合基因调控网络来捕获细胞内过程。
生成真实数据多年来一直是一个重大挑战:为了解决这个问题,正在开发真实交互的精选数据库,以帮助基准测试和调整计算工具。此外,努力生成金标准数据集对于通过适当的指标(如准确性、精密度、召回率、f分数和均方根误差)评估工具性能至关重要。建立此类参考数据的尝试通常涉及从文献中手动整理CCIs,包括不同的细胞类型和分子介质,如CITEdb和Cellinker所示。
包括配体和受体的亚细胞定位可完善对相互作用细胞的预测:例如考虑到膜蛋白相互作用的生物物理特征(如定位、强度和细胞骨架耦合)的新算法可以用于研究免疫突触和胚胎发育的动力学。
大多数实验方法仅限于原位和体外部署,在体内开展的工作很少:预计将出现更多的技术来报告更多经过验证的CCIs,并将其与下游测序和计算分析相结合。例如ullipstic可以与scRNA-seq结合来解开复杂的CCIs;为了帮助扩大实验获得的CCIs和LRIs网络,实验方法可以与多重蛋白质成像方法(如CODEX)相结合,这是一种使用抗体与DNA条形码结合的方法,可以在空间上可视化组织内的多种蛋白质标记物。此外,新兴的标记分泌信号的方法[和远程通信机制对于使这些高通量实验方法在跟踪CCIs时更加全面至关重要。
//
建议对技术细节感兴趣的小伙伴请参考文献原文~
对于文献整理过程中有翻译不当或错误也欢迎大家在评论区留言指出,互相交流学习!
参考文献
Armingol, E., Baghdassarian, H.M. & Lewis, N.E. The diversification of methods for studying cell–cell interactions and communication. Nat Rev Genet (2024). https://doi.org/10.1038/s41576-023-00685-8