最近发现了一个专门收集整理了肿瘤转移数据的数据库工具——HCMDB,网址:http://hcmdb.i-sanger.com/index。该工具由华东师范大学的董东教授于2017年发表于著名的《Nucleic Acids Research》,影响因子11.147分。

这个神器收集了GEO和TCGA上29种肿瘤,45种肿瘤亚型,38个转移部位,共124个数据集和7081篇文献的信息。其中表达数据涵盖了mRNA、miRNA、lncRNA, 真香!
下面我给大家简单介绍一下网站的使用,网站的主页如下:可以直接在“Quick Search”直接搜索你想要找的基因。
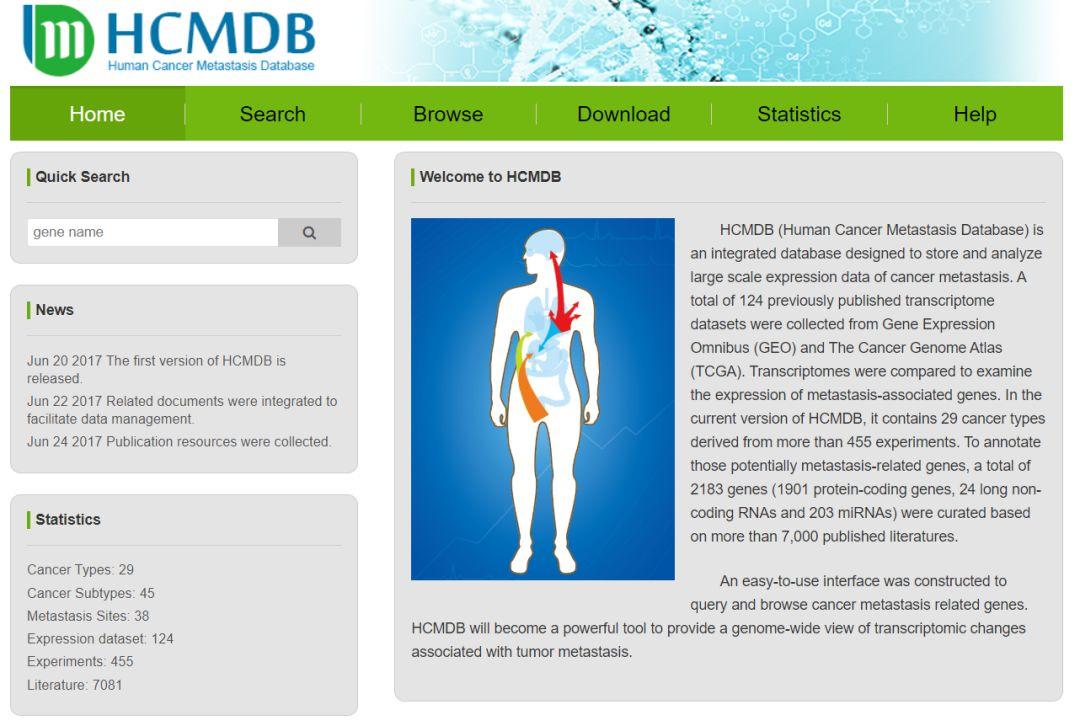
或者在绿色的“Search”栏里放入你要搜索的内容,可以混合搜索不同类型的RNA。也有多种格式的基因名供你选择,包括ENSEMBL,RefSeq,NCBI Entrez gene ID,uniprot ID。然后点击“Submit”

然后在搜索结果界面会展示上面搜索内容的结果,整个页面每次只会展示一个RNA,点击哪个字体加粗,然后在后面展示出来。我们以“CDKN2A”为例。

网页会展示基因的概览

先前研究对这个基因参与肿瘤转移的描述(这个真是好,简单直接的文献证据)

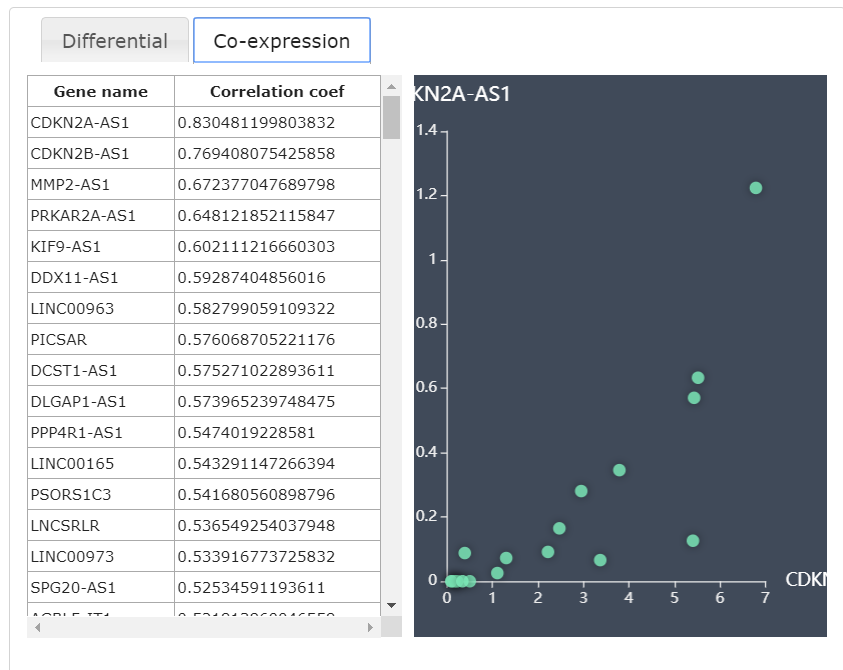
接下来展示了这个基因在不同研究中的表达。点击“Cancer Name”的栏目可以选择你要研究的肿瘤,点击“Filter”后开始筛选。图中的“DIFF”表示有差异表达,“NOT”当然就是不显著了,点击那个图标,可以跳出差异表达的box图,还有共表达的结果。
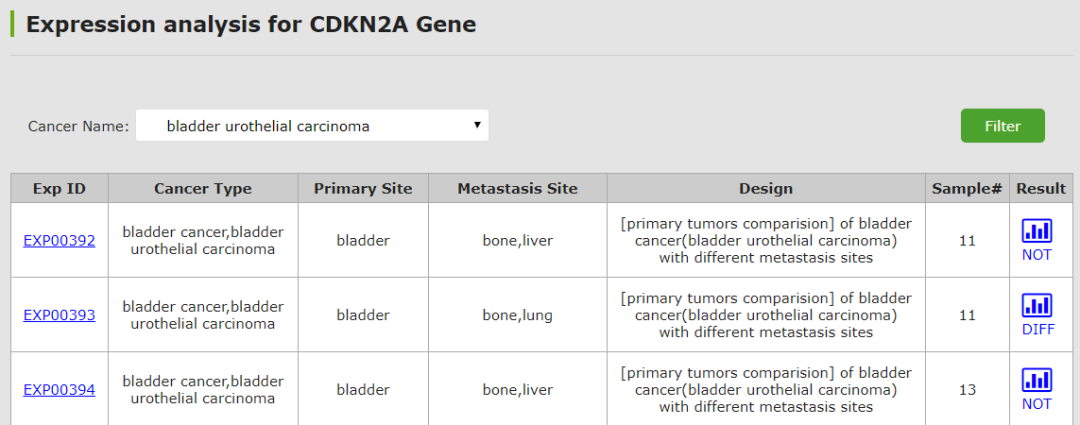

点击“Exp ID”栏下的蓝色字体可以跳转到对单项研究的结果展示部分:
先是会展示一个概览
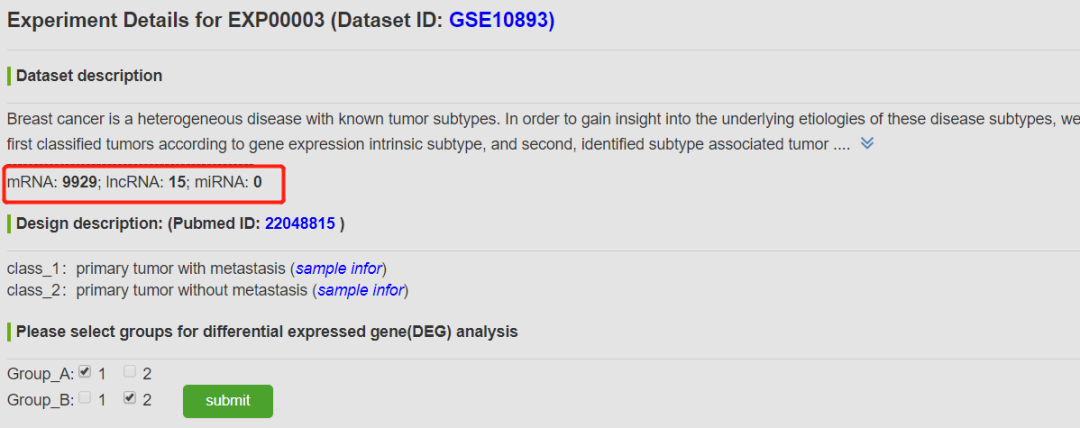
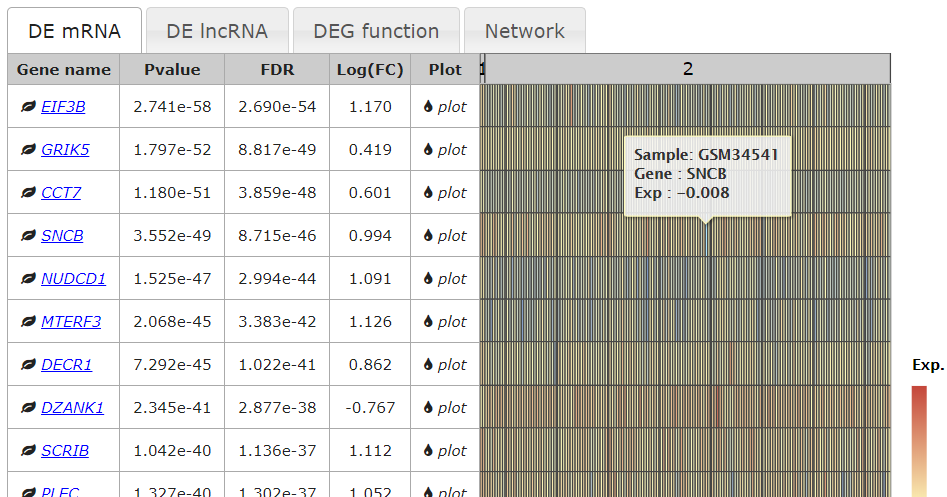
然后会展示该研究的差异表达内容,包括pvalue,FDR,log(FC)和热图。
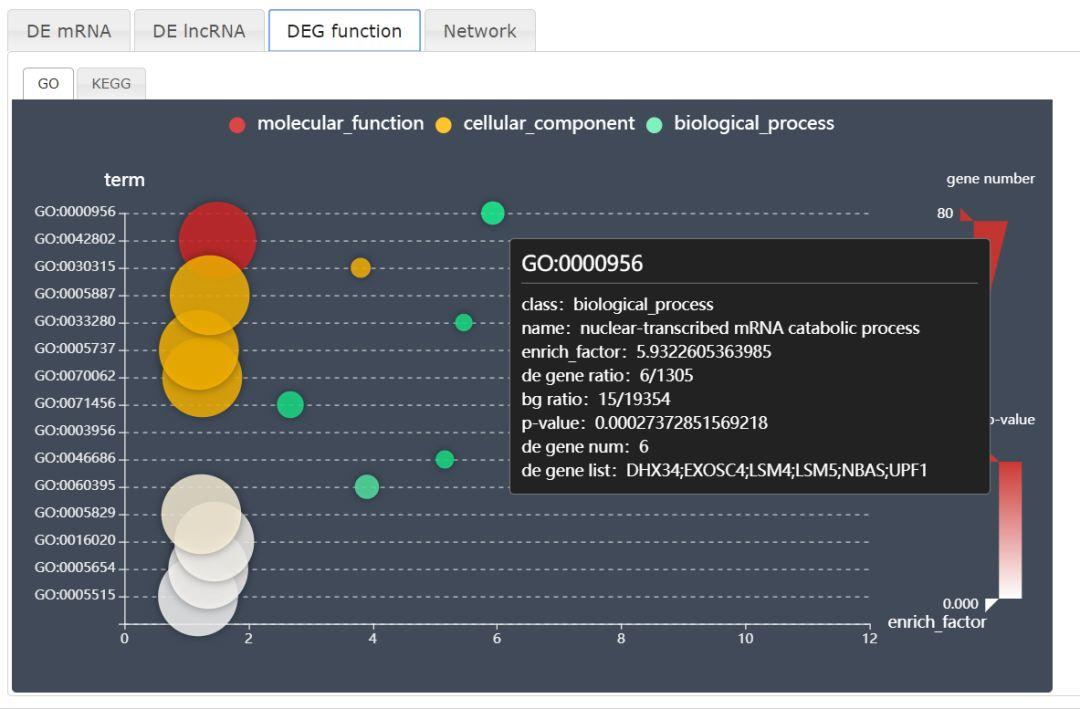
点击DEG_function,会展示该研究里的差异表达基因富集到的通路,把鼠标放在圈圈上,可以展示通路的详细信息,包括通路的分类、名称、富集分数、pvalue等。
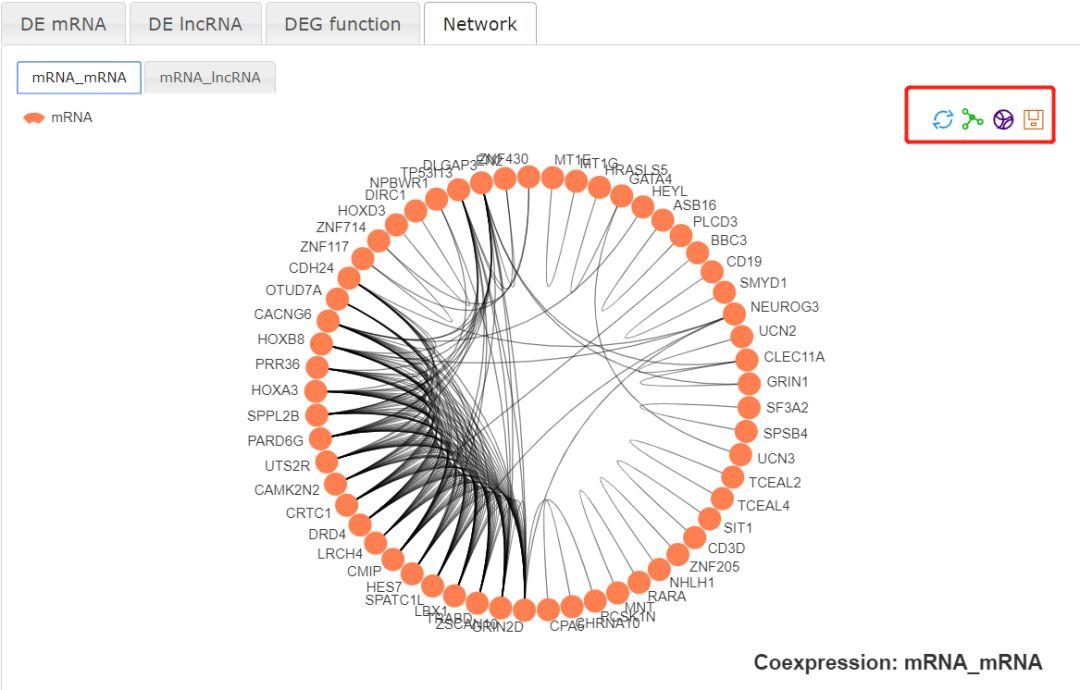
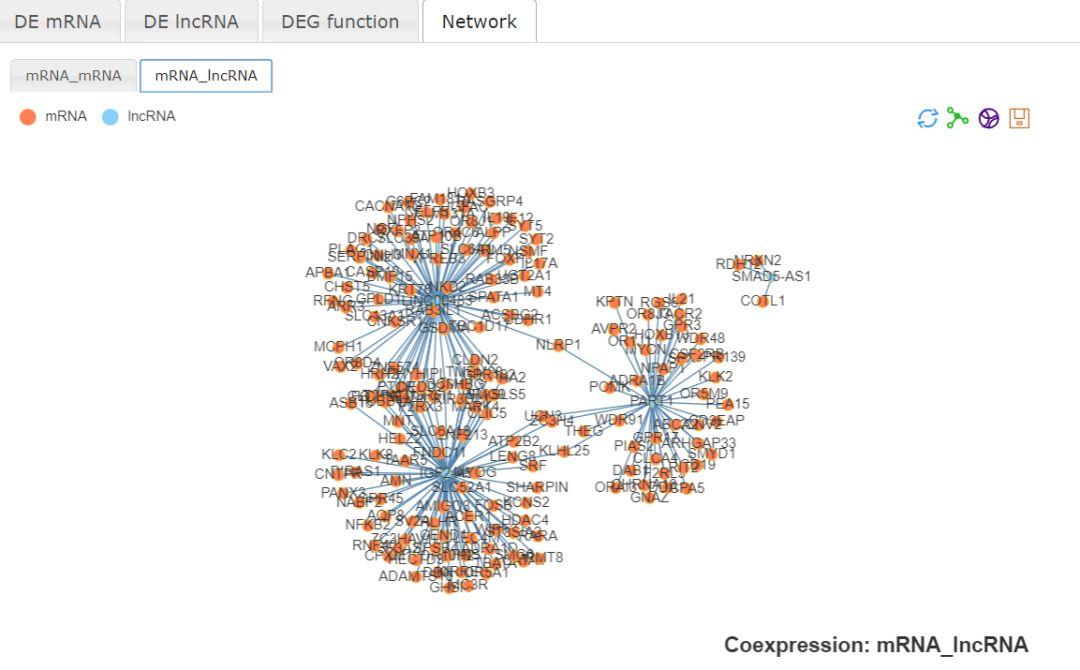
然后可以在Network部分可以展示差异表达基因的网络图,包括mRNA-mRNA和mRNA-lncRNA两种,点击右上角的图标可以展示不同的展示风格。也可以保存图片。
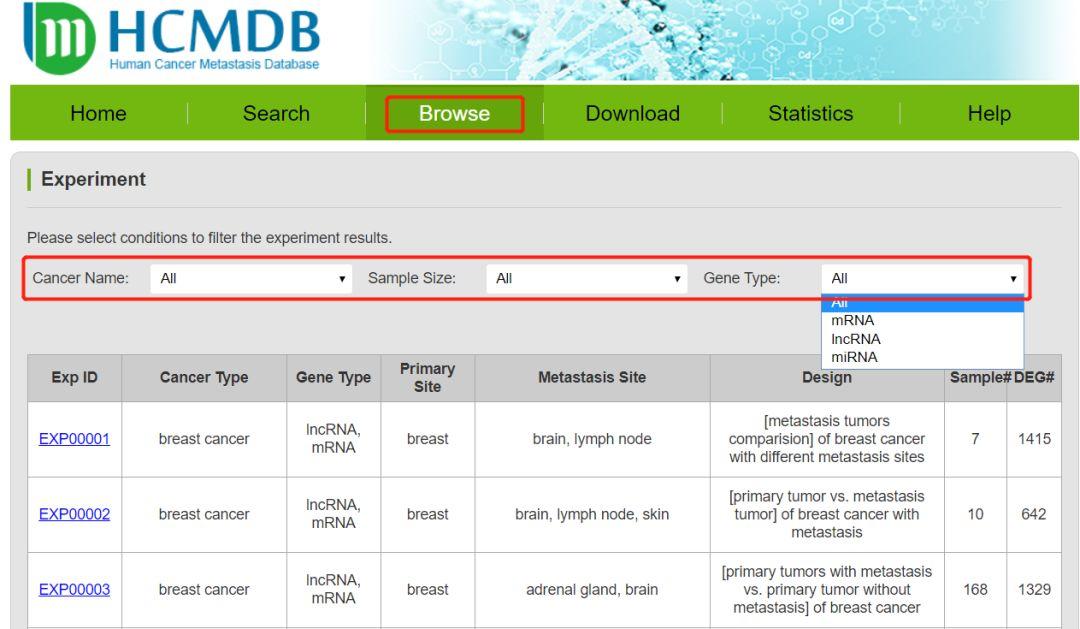
除了根据基因进行筛选,还可以在Browse开启你的探索之路,选择研究的肿瘤、过滤样本数、选择RNA类型,这些都一气呵成。当然,研究肿瘤的时候常常也会重点关注一些特殊的转移部位, 比如肠癌的肝转移。
工具的介绍就到这里了,这个工具的数据量真的非常可以了,使用起来对用户也比较友好,笔者花了10分钟就基本搞清楚用法了。
工欲善其事必先利其器,祝大家都能利用好这个工具,发出好文章!