今天的是三周合计15天的数据挖掘授课学员一点一滴整理的授课知识点笔记哦,还有互动练习题哈,欢迎大家点击文末的阅读原文去关注我们学员的公众号哦!
# Group(实验分组)和ids(探针注释)rm(list = ls()) load(file = "step1output.Rdata")library(stringr)# 标准流程代码是二分组,多分组数据的分析后面另讲# 生成Group向量的三种常规方法,三选一,选谁就把第几个逻辑值写成T,另外两个为F。如果三种办法都不适用,可以继续往后写else ifif(F){ # 1.Group---- # 第一种方法,有现成的可以用来分组的列 Group = pd$`disease state:ch1` #现成分组,要判断是哪一列}else if(F){ # 第二种方法,自己生成 Group = c(rep("RA",times=13), rep("control",times=9)) Group = rep(c("RA","control"),times = c(13,9))}else if(T){ # 第三种方法,使用字符串处理的函数获取分组 Group=ifelse(str_detect(pd$source_name_ch1,"control"), "control", "RA")}#想要使用哪种方法,就把哪个方法改成T,其余的改成F# 需要把Group转换成因子,并设置参考水平,指定levels,对照组在前,处理组在后#因子:见下文Group = factor(Group,levels = c("control","RA"))Group #2.探针注释的获取-----------------#探针ID和基因symbol的对应关系#捷径library(tinyarray)find_anno(gpl_number) #打出找注释的代码ids <- AnnoProbe::idmap('GPL570').db#四种方法,方法1里找不到就从方法2找,以此类推。#灰色部分需要替换#方法1 BioconductorR包(最常用)gpl_number#http://www.bio-info-trainee.com/1399.html 进入这个网站,网页搜索GPL编号,可以定位到对应R包,要加.db。if(!require(hgu133plus2.db))BiocManager::install("hgu133plus2.db")#安装library(hgu133plus2.db)#加载ls("package:hgu133plus2.db")#看这个R包中有那些数据ids <- toTable(hgu133plus2SYMBOL)#提取R包中有用的信息,tablehead(ids)# 方法2 读取GPL网页的表格文件,按列取子集##https://www.ncbi.nlm.nih.gov/geo/query/acc.cgi?acc=GPL570if(F){ #注:表格读取参数、文件列名不统一,活学活用,有的表格里没有symbol列,也有的GPL平台没有提供注释表格 b = read.delim("GPL570-55999.txt",#这个文件在上面网站上下载,放在工作目录里 check.names = F, comment.char = "#") colnames(b) ids2 = b[,c("ID","Gene Symbol")] colnames(ids2) = c("probe_id","symbol") k1 = ids2$symbol!="";table(k1) k2 = !str_detect(ids2$symbol,"///");table(k2) ids2 = ids2[ k1 & k2,] # ids = ids2#如果不用修改上面的内容,就直接ids=ids2} # 方法3 官网下载注释文件并读取##http://www.affymetrix.com/support/technical/byproduct.affx?product=hg-u133-plus# 方法4 自主注释#https://mp.weixin.qq.com/s/mrtjpN8yDKUdCSvSUuUwcAsave(exp,Group,ids,gse_number,file = "step2output.Rdata") |
|---|
因子: Group factor(Group)
#.变成因子后没有引号 #水平 因子里面的取值,顺序重要,第一个位置上的是参考水平 # 应该让对照组在前,处理组在后,保证差异分析不反 factor(Group) #水平按照首字母顺序排序 factor(Group,levels = c("control","RA")) #水平按照代码里写的顺序排

找注释代码的方法
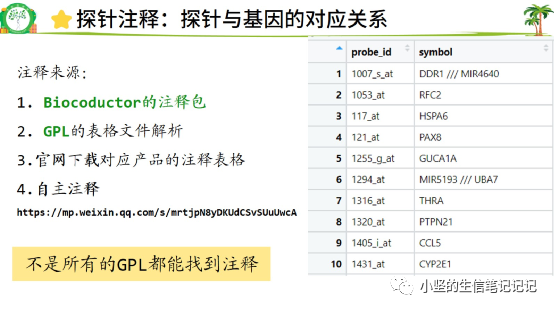
方法一

方法二 http://www.bio-info-trainee.com/1399.html
方法三 官网下载注释文件并读取
##http://www.affymetrix.com/support/technical/byproduct.affx?product=hg-u133-plus
有可能GeneSymbol会是其他ID,需要转换

一个探针可能代表不止一个基因(如上)可以去掉。也有可能不对应基因symbol,转换方法如下:K1去掉空格,k2去掉///

有可能只有序列(如下),需要自主注释
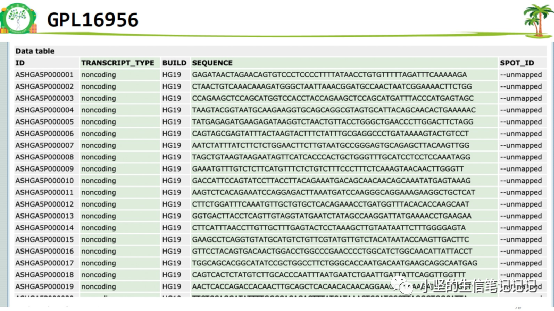

方法四 自主注释
#https://mp.weixin.qq.com/s/mrtjpN8yDKUdCSvSUuUwcA
save(exp,Group,ids,gse_number,file = "step2output.Rdata")